VariantGrid
VariantGrid est un outil d'interprétation de variations génomiques développé en Python via Django par les australiens de l'ACRF Cancer Genomics Facility sous Business Source License 1.1, davantage orienté vers le partage de résultats d'interprétation que pour le diagnostic pur.
Note
Pour tester l'outil, RDV à ce lien
et utilisez :
Username : match
Password : 2J|?u=9k}5_hQ7LTZ/Fk
Présentation générale
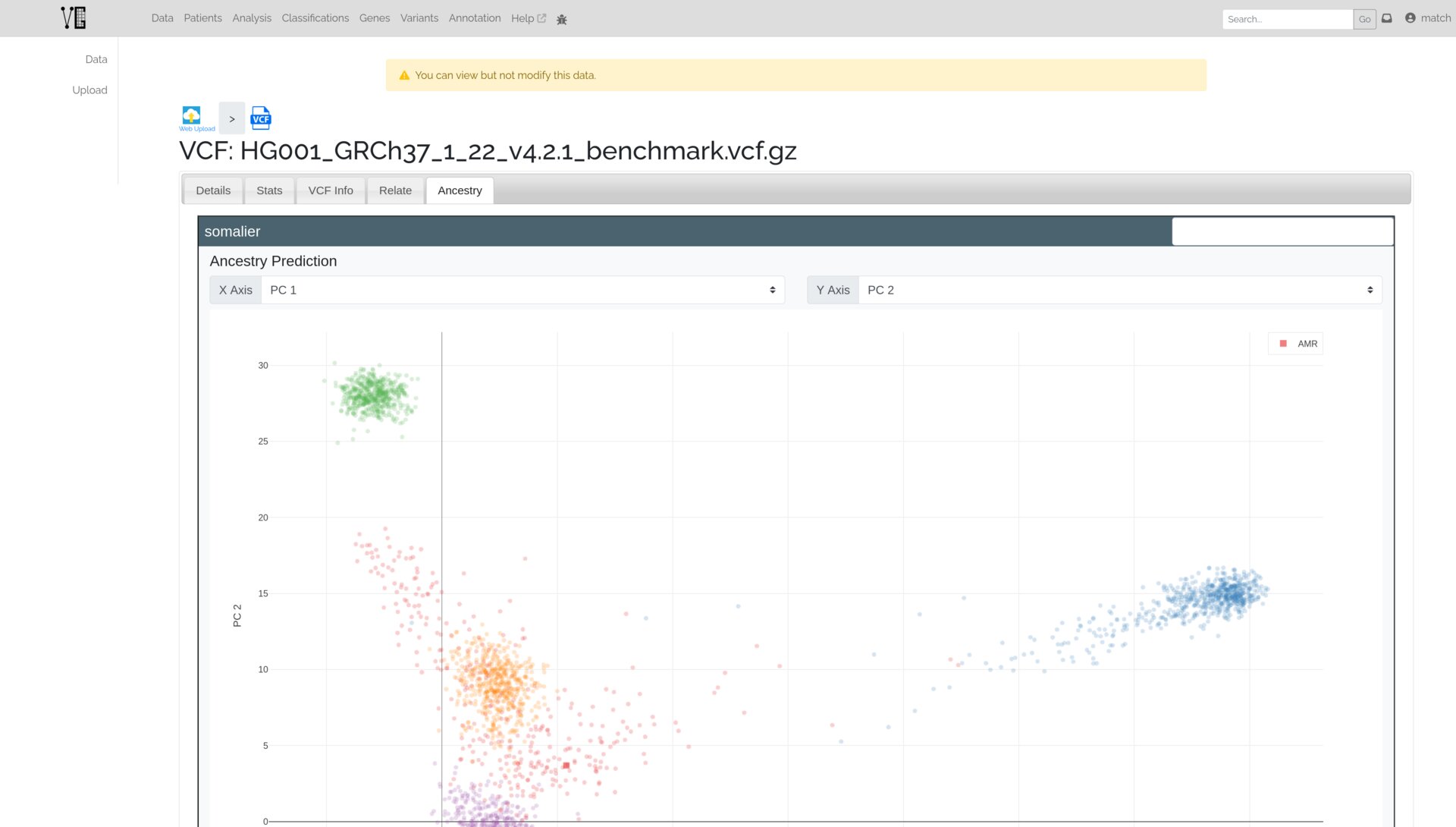
Annotations
Leur poster offre une présentation générale du projet et un aperçu du fonctionnement de l'outil.
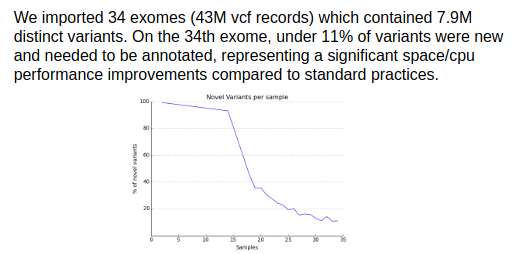
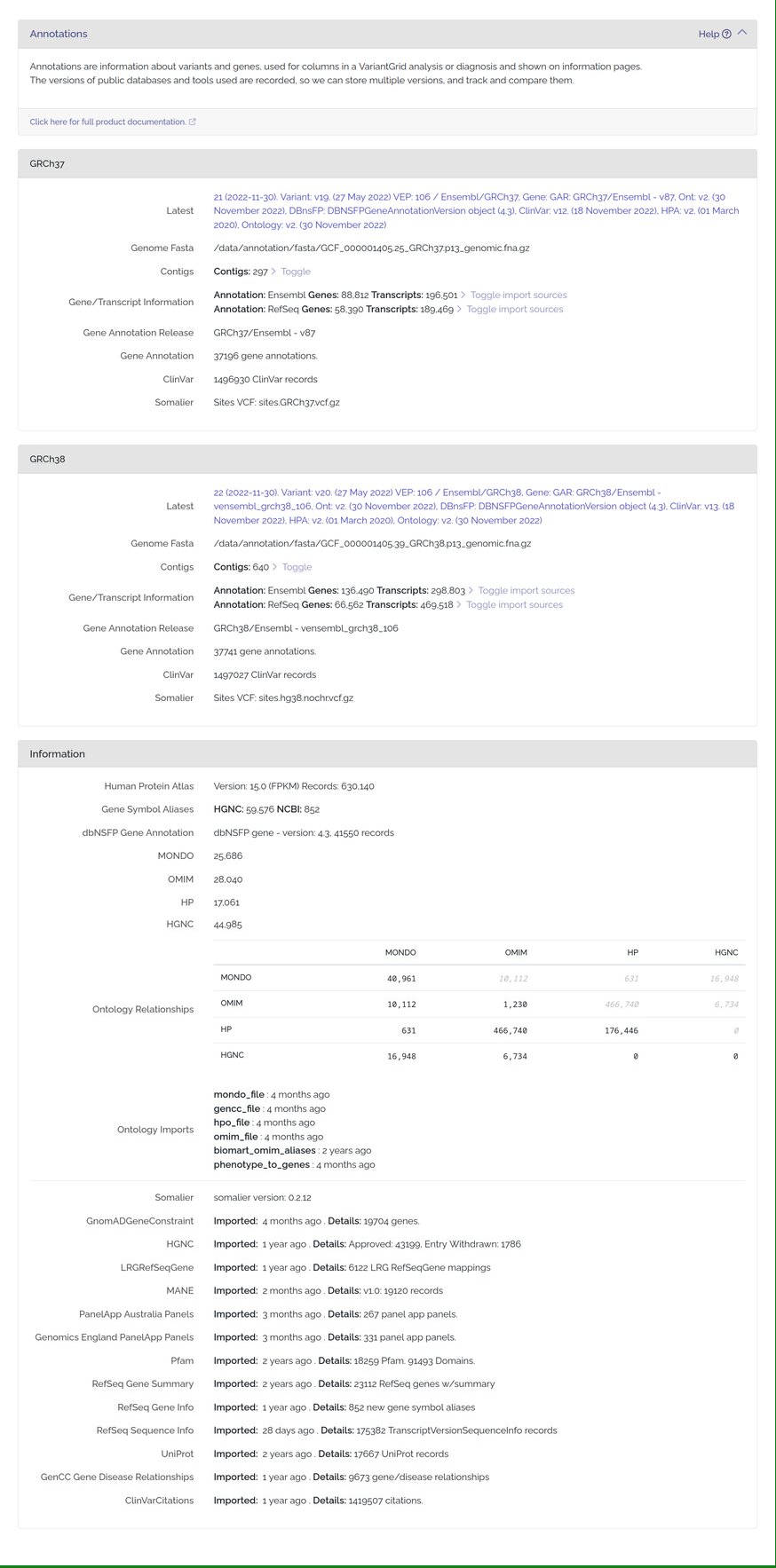
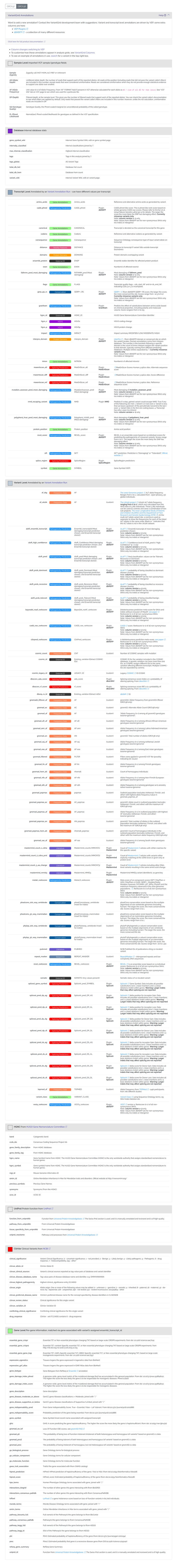
Les VCF chargés sont intégralement annotés par VEP via une configuration globale et choisie par les admins. Il est possible via une ligne de commande d'ajouter de nouveaux champs à récupérer des annotations de VEP ce qui entraînera un ajout de colonnes dans la DB.
Les annotations sont versionnées, évitant une ré-annotation ultérieure de variation déjà connues.
De leur aveu même, ce fonctionnement n'est cependant pas compatible avec l'analyse de génomes ou au delà d'une masse critique d'exomes.
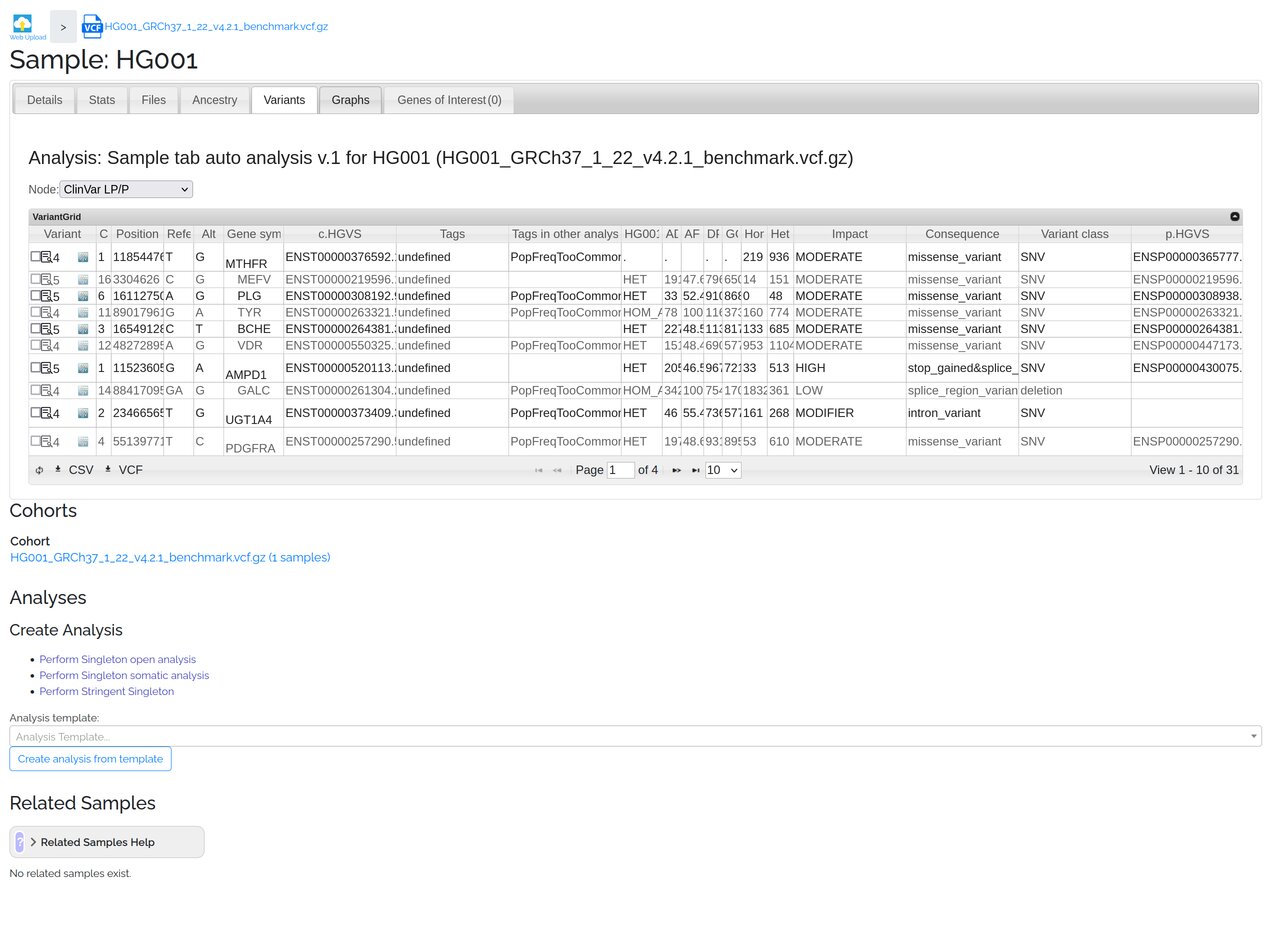
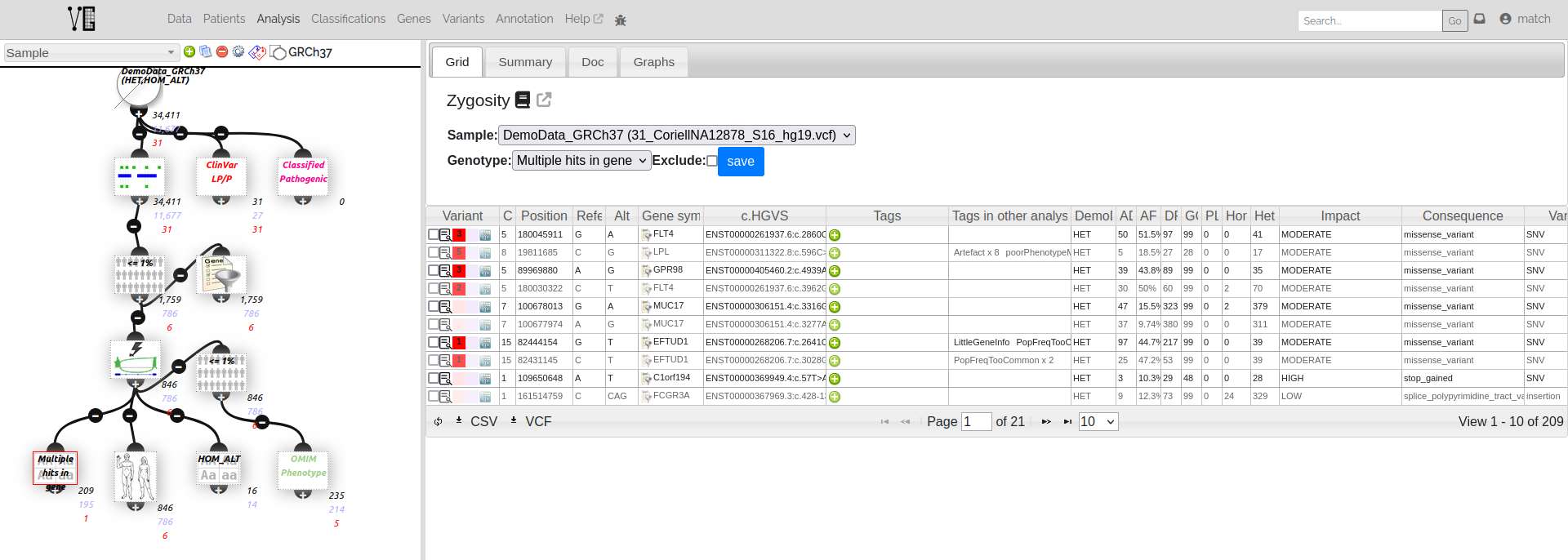
Filtrage
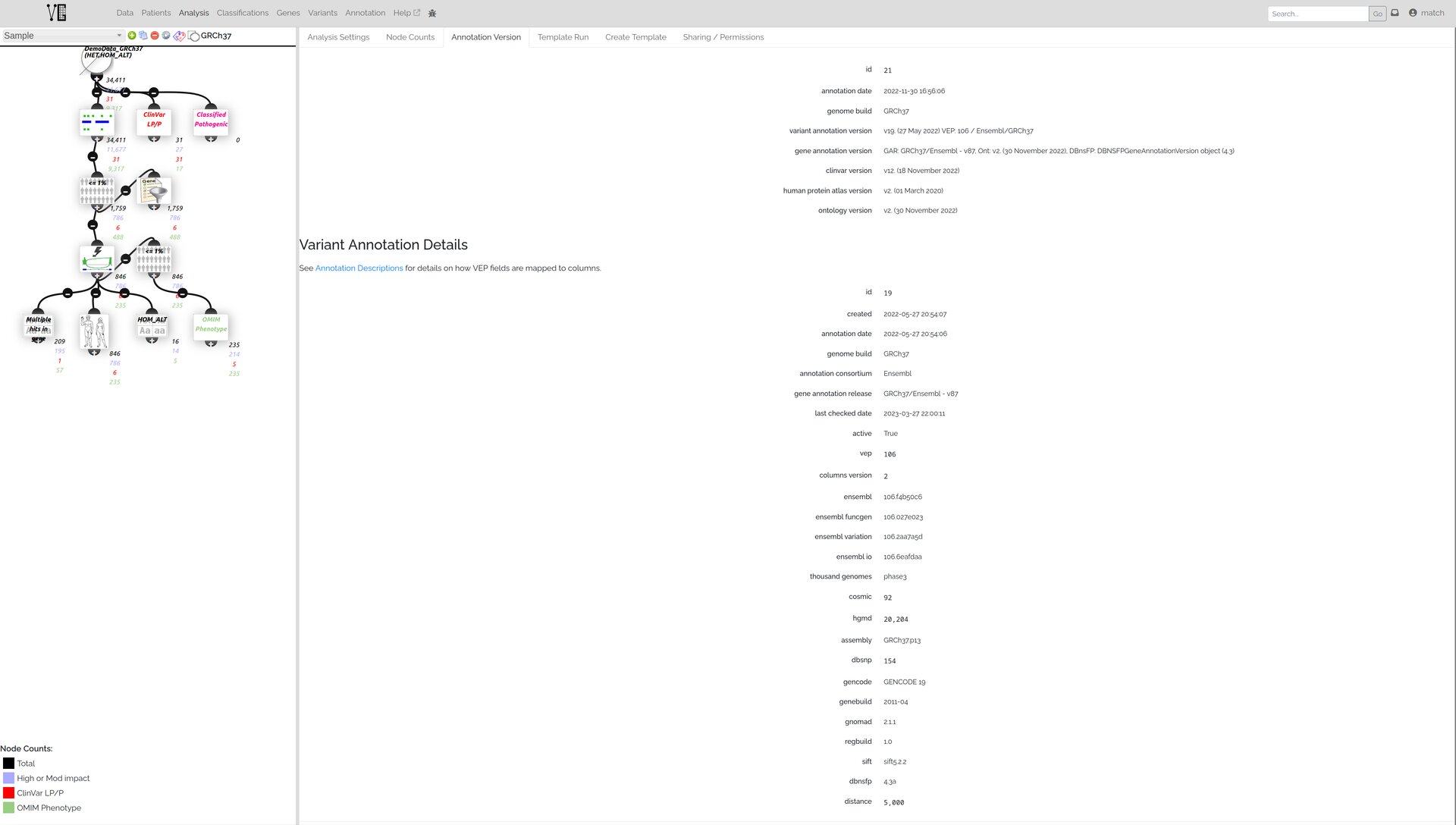
Un point intéressant de cet outil est son application de filtres via un graphe rappelant sommairement le fonctionnement d'Alissa. L'UX est tout à fait perfectible, notamment dans la création du filtre, mais offre d'ores et déjà une représentation visuelle, facile à traverser lors d'une analyse, réutilisable avec un suivi de version et partageable entre utilisateurs selon les autorisations accordées par son créateur.
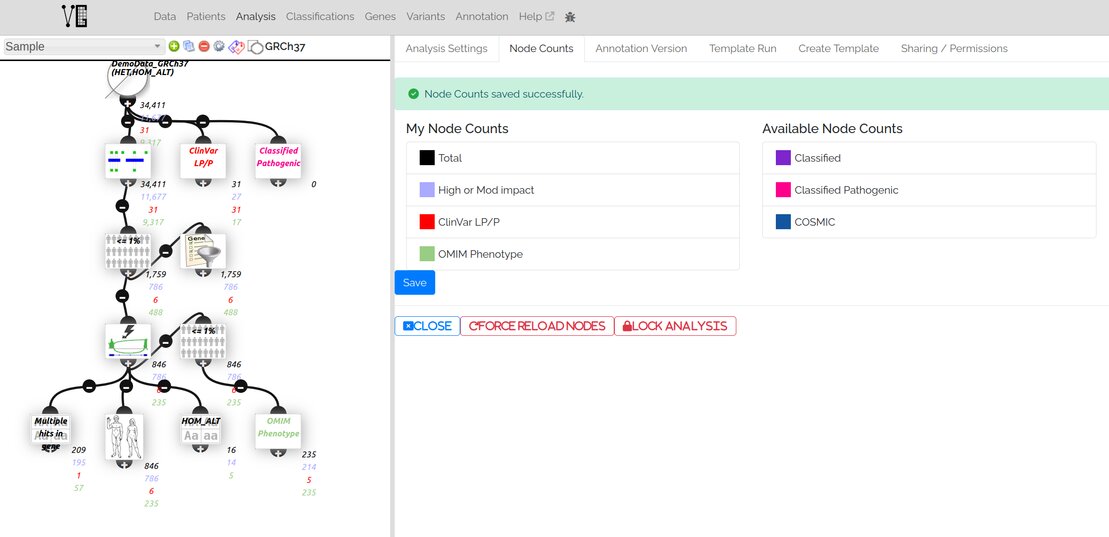
En prenant l'example du graphe ci-dessous (Analyses/Node count), le recomptage en 4
sous-sets des variations de chaque nœud prend moins de 5s pour un sample de 34411
variations et environ 2min40 pour 3,9M variations. Ce comptage est fait de manière
progressive, permettant de commencer à fouiller les premiers nœuds avant la fin du
comptage du graphe entier.
Ce comptage est conservé lors d'une réouverture ultérieure de l'analyse.
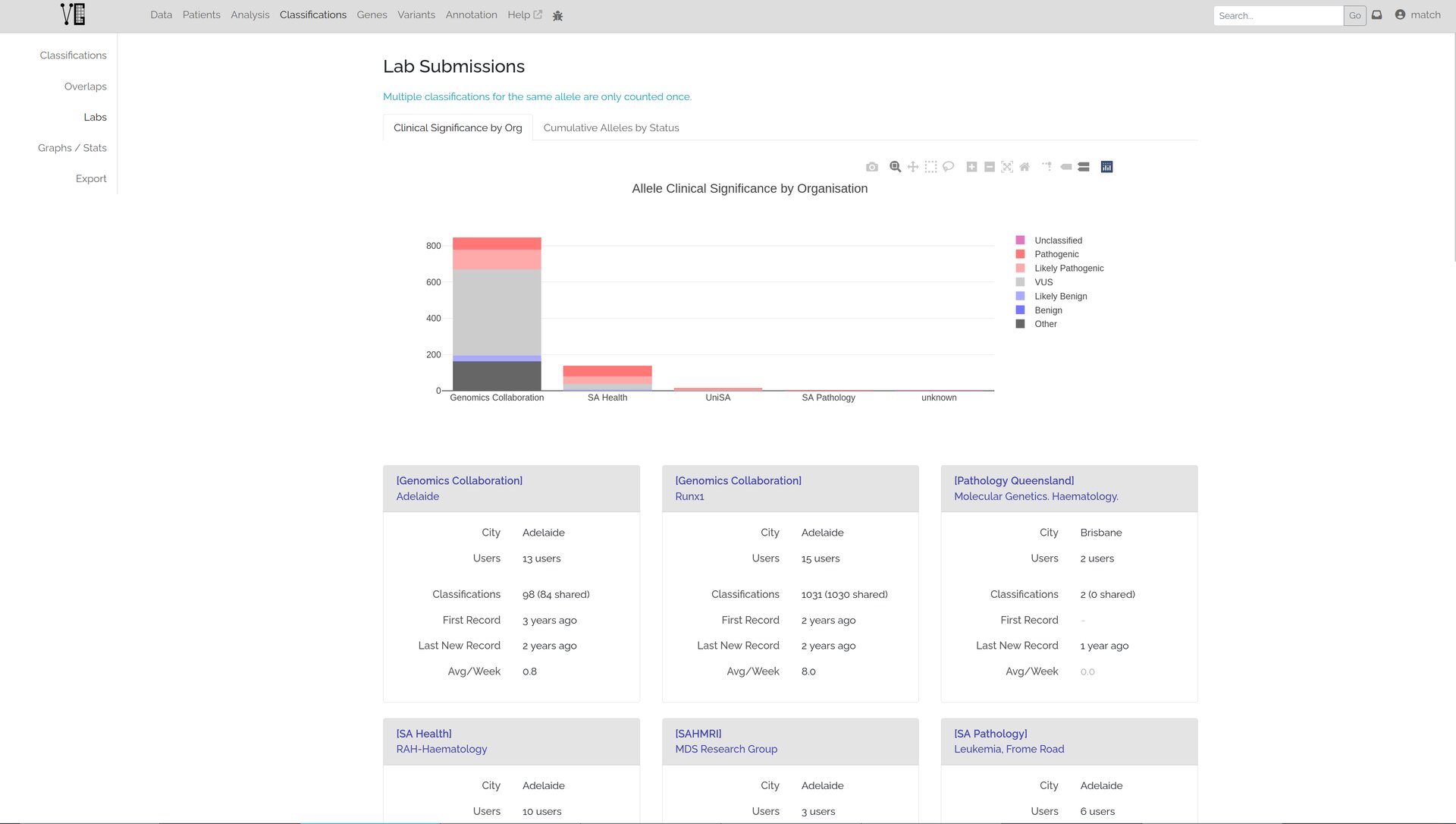
Partage
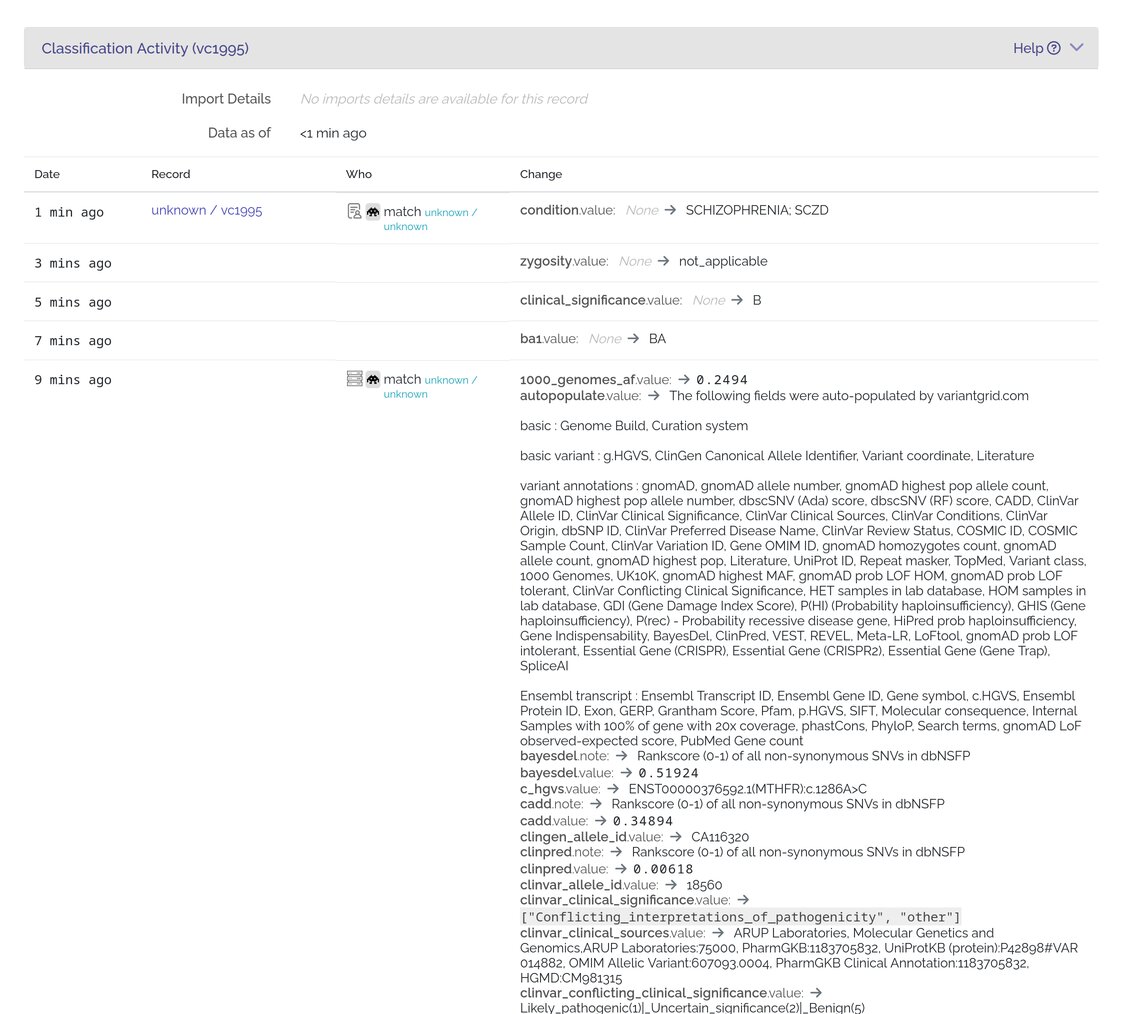
Cet outil reste orienté avant tout vers le partage de résultats plutôt que le diagnostic et a donc un fort investissement dans les échanges entre laboratoires et un upload simplifié de résultats sur des outils comme ClinVar et GeneMatcher.
Interface
Avis
| Points positifs | Points négatifs |
|---|---|
| Repose sur Django, permettant de comparer des solutions | … mais ont une approche foncièrement différente |
| Le graphe de filtres | … bien que largement perfectible dans son utilisation en front |
| La reconnaissance de termes dans les dossiers patients | … bien que perfectible côté front |
| Une annotation versionnée censée faire gagner du temps au fil des ajouts | … mais menant ineluctablement à des problèmes de scalabilité |
| Un form de classification plutôt rodé | Un fonctionnement pas comparable dû à la législation européenne |
| Customisation user du tableau et partage de ces configs |