Scout
Scout est un outil d'interprétation de variations génétiques sous licence BSD-3 en
développement encore actif depuis au moins 4 ans. L'outil est codé en Python et repose
sur Flask.
Sa philosophie est davantage orientée vers un partage de découvertes entre laboratoires
qu'offrir un outil d'aide au diagnostic internalisé.
Note
Il est possible d'installer facilement
une instance de démo. Pour s'y connecter,
l'email à entrer est clark.kent@mail.com
Présentation générale
- Découpage en Institutes dans lesquels sont inclus des cases.
- Gère SNV, indels, SV et STR pour les maladies rares et la somatique.
- Orienté diag et interprétation de variations
- Partage sur ClinVar intégré
Présentation détaillée
L'écran d'accueil nous liste les projets disponibles.
Accueil
L'écran principal permet d'accéder à :
- la liste des Institutes (idée approchante de nos projets) pour accéder aux cases
- la liste des gènes dans la DB permettant d'accéder à une vue détaillée de ceux-ci
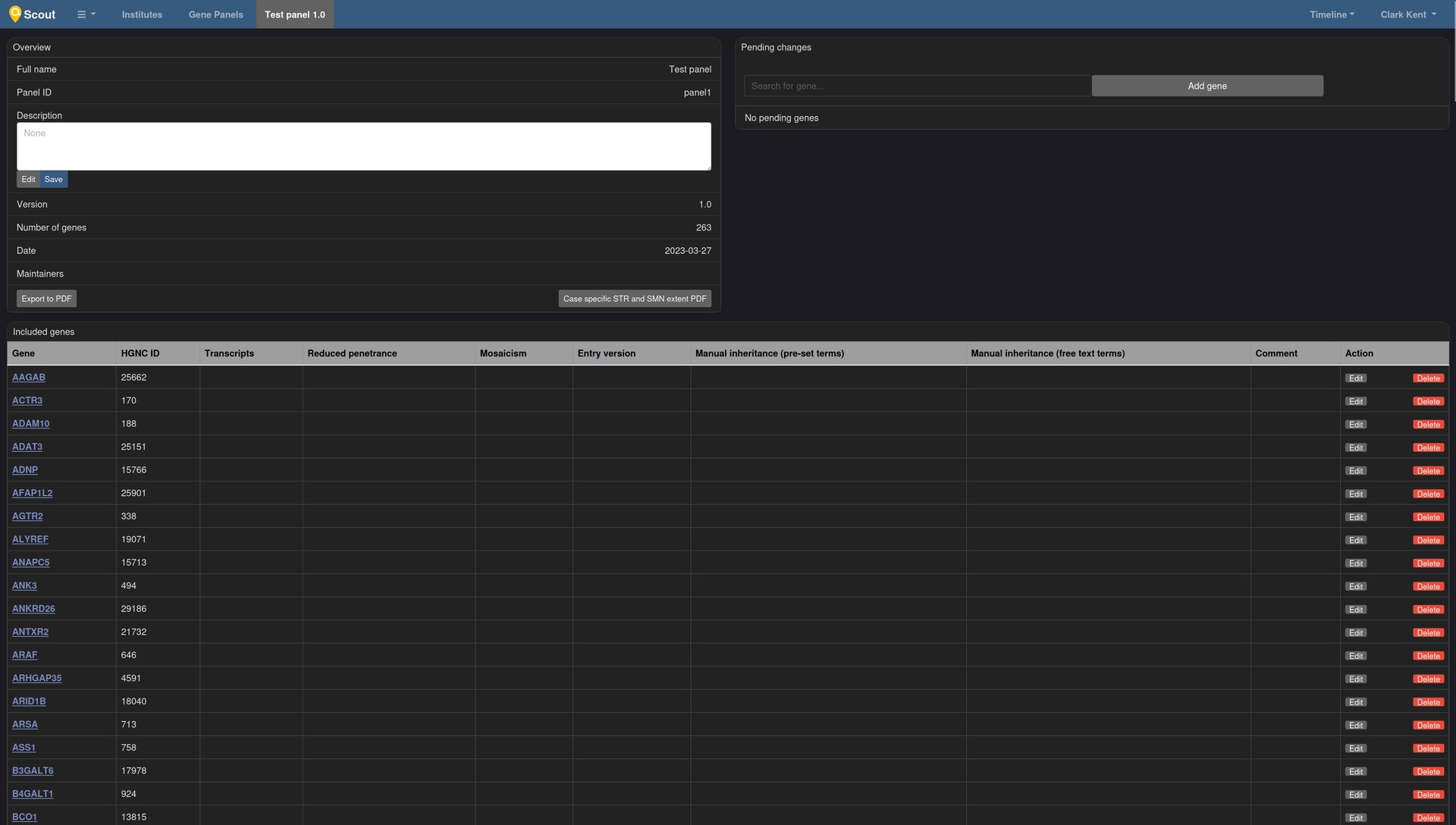
- la liste des panels de gènes dans la DB et création de nouveaux
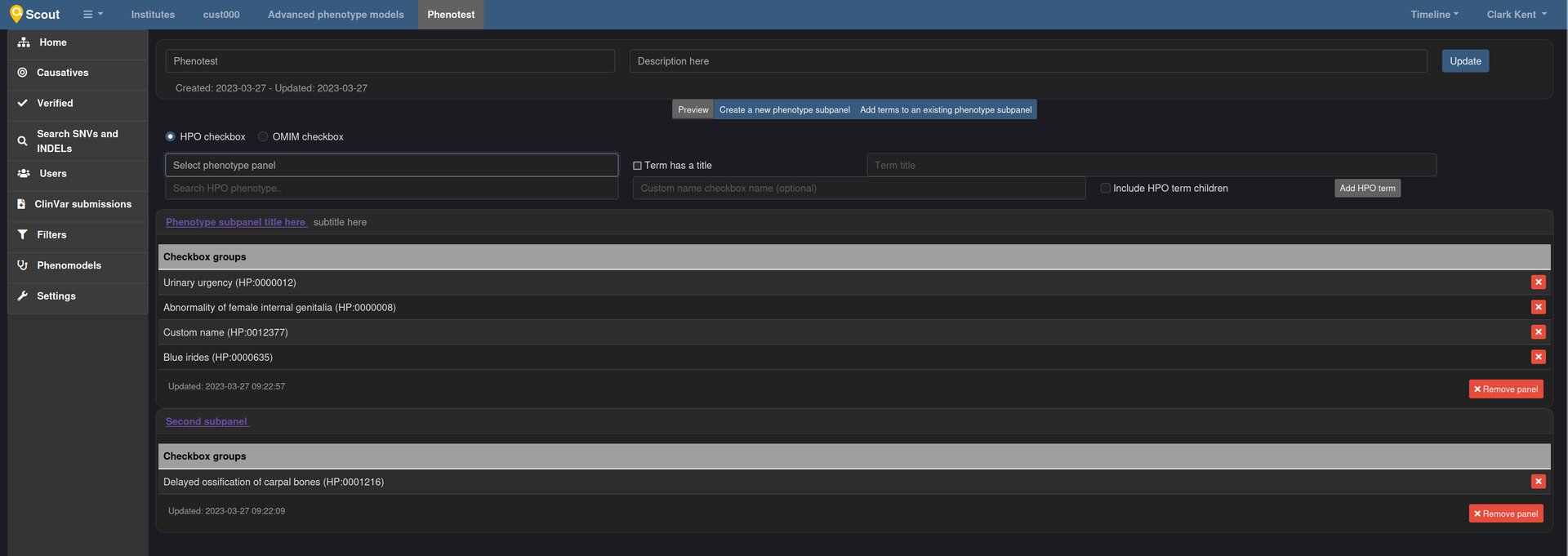
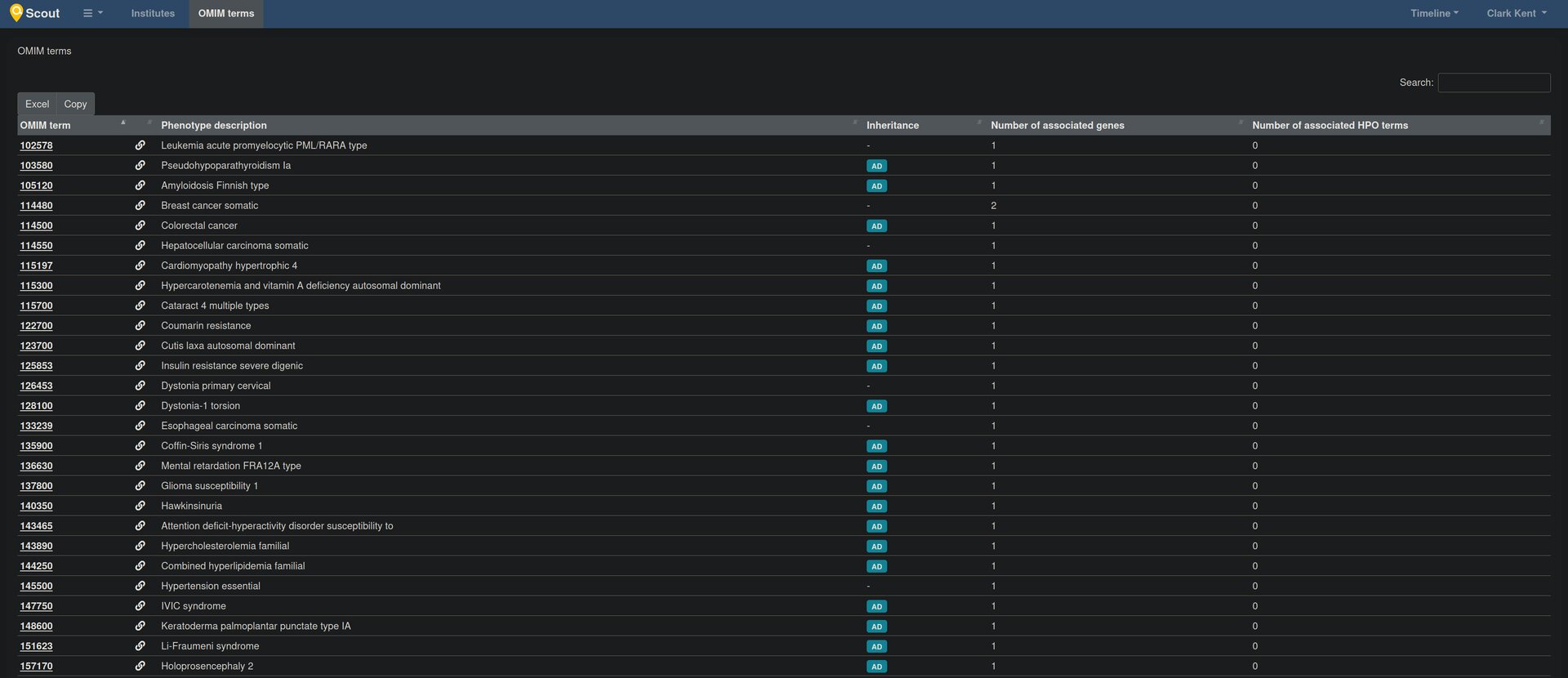
- la liste des termes HPO
- la liste des termes OMIM
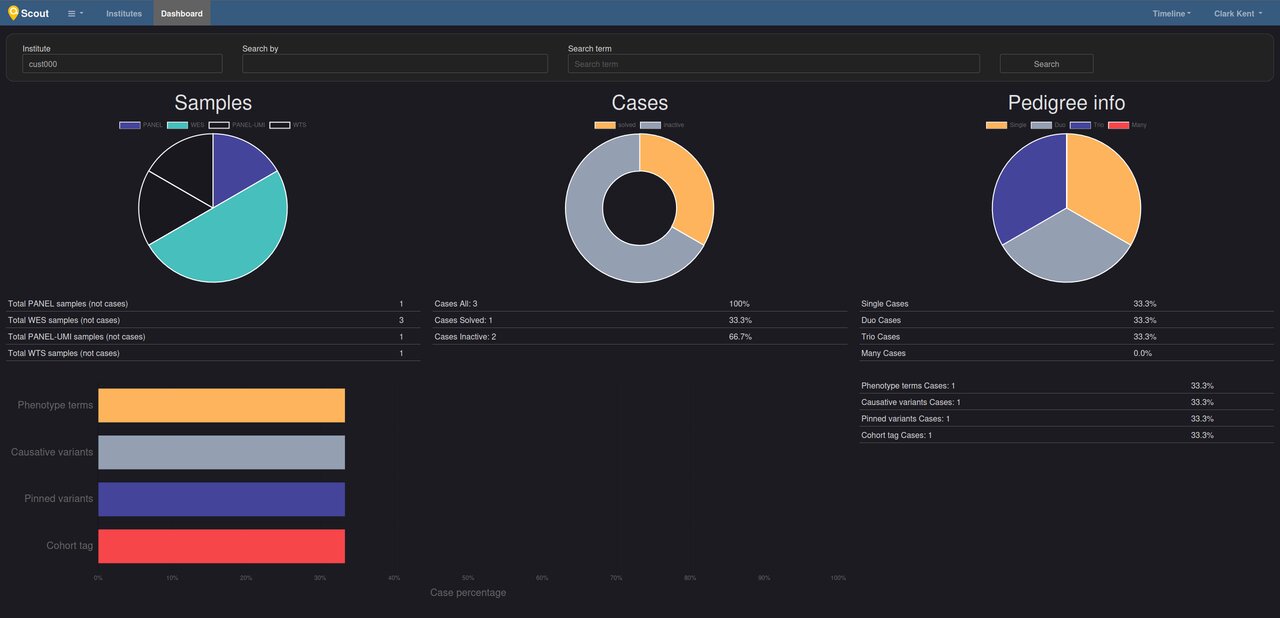
- un dashboard de suivi de l'instance et des Institutes
Institutes
- Permet de réunir les cases en attente d'interprétation, en cours, résolus et ignorés.
- Permet de retrouver facilement les variations retenues causales au sein d'un Institute.
- Permet de définir des droits d'accès pour les utilisateurs.
- Permet de partager des filtres.
- Permet de créer des panels de phénotypes HPO.
Cases
Ce sont l'équivalent des interprétations pour Diagho incluant l'écran d'accueil et les
tableaux pour les différents types de données.
Ne contient pas de page dédiée aux QC mais génère automatiquement des Reports que ce
soit sous un format interne pour avoir tout le débrief d'une interprétation comme sous
la forme d'un compte-rendu d'analyse officiel.
Les tableaux d'interprétation quant à eux sont présentés en dur avec un set de filtres
limités mais partageables.
Interface
Cliquer sur un id renvoir vers https://hpo.jax.org/app/browse/term/HP:<id>
Cliquer sur le chaînon renvoi vers une vue détaillée interne listant les symboles de gènes associés, le type de transmission et les autres phénotypes associés.

Cliquer sur un id renvoir vers https://omim.org/entry/<id>
Cliquer sur le chaînon renvoi vers une vue détaillée interne listant les symboles de gènes associés, le type de transmission et les autres phénotypes associés.
Écran à l'ouverture d'un cas. Équivalent de l'onglet Général prévu pour les interprétations sur Diagho.
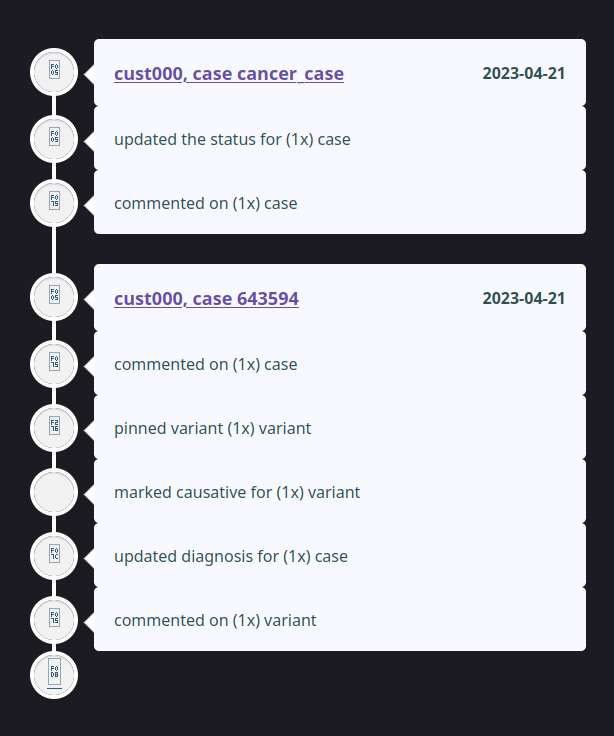
Offre un résumé du cas, des individus associés, du tableau clinique. Contient un audit trail de toute action liée à ce cas et permet d'ajouter des commentaires signés et datés partagés aux autres utilisateurs.
Les boutons en haut (Clinical SND and INDELs…) renvoient vers les vues détaillées dans Tableau.

Écran de base à l'ouverture d'un type de donnée. Les colonnes sont fixes et non configurables par l'utilisateur. La position exacte n'est même pas précisée sans rentrer dans Vues détaillées/Variation
Un tri des variations par priorisation sur rank score est effectué (technique reposant toujours sur des choix arbitraires et pouvant tout autant faire gagner du temps que rendre le diagnostic impossible).
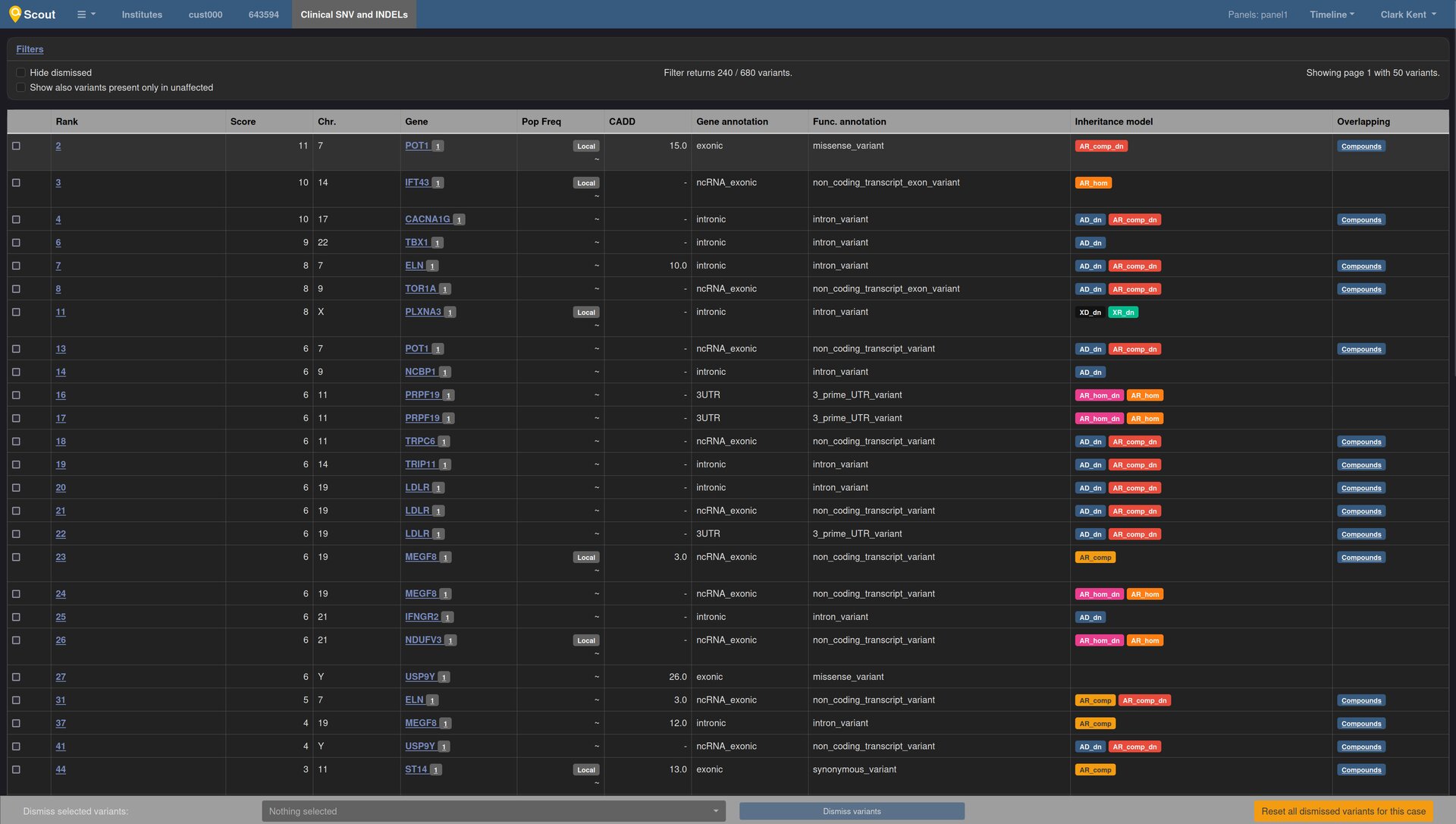
Filtre par GT
 Filtre par région touchée
Filtre par région touchée
 Pathogénicité estimée sur ClinVar
Pathogénicité estimée sur ClinVar
 Modèle génétique (estimé par Scout)
Modèle génétique (estimé par Scout)

Tooltip des gènes
 Tootip des fréquences
Tootip des fréquences
 Tooltip d'hétérozygotie composite détectée
Tooltip d'hétérozygotie composite détectée
 Tooltip en cas de présence d'un SV couvrant cette position
Tooltip en cas de présence d'un SV couvrant cette position
 Choix pour marquer une variation comme non pertinente
Choix pour marquer une variation comme non pertinente
 Tooltip résultante
Tooltip résultante

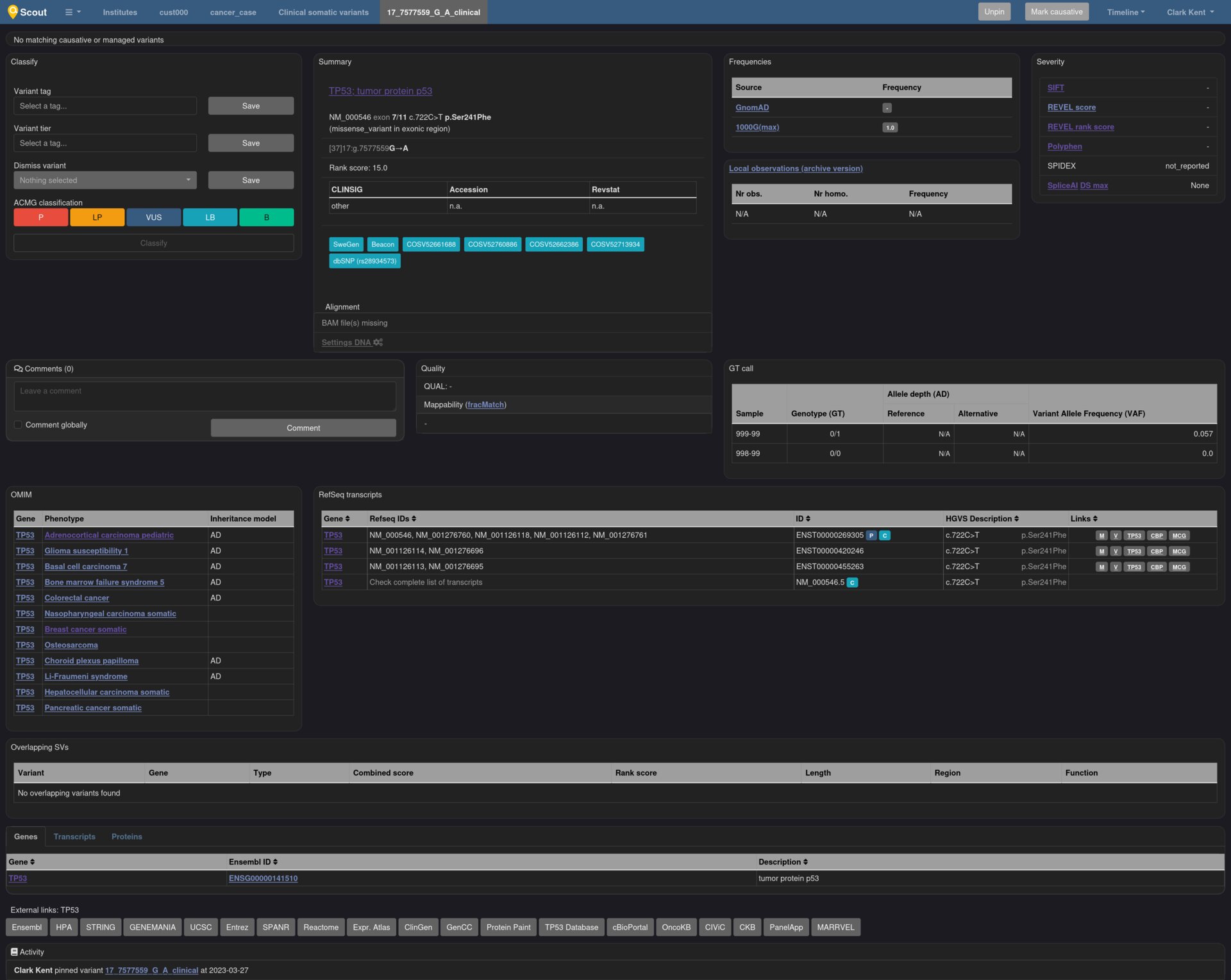
Vue détaillée d'une variation dans le cadre d'une interprétation.
Possibilité d'ajouter un commentaire signé, daté et partagé ou non aux autres utilisateurs.
Contient des liens directs vers un grand nombre de BDD, liées à la variation ou au gène.
Audit trail lié à la variation et permettant de garder en mémoire toute action utilisateur à son sujet.

Avis
| Points positifs | Points négatifs |
|---|---|
| Beaucoup de bonnes idées | … mais pas toujours pleinement implémentées |
| Tri priorisation possible | … mais appliqué de force |
| Gestion des HPO et OMIM | … mais intégration semble-t-il encore à faire pour être utilisés en interprétation |
| Bonne traçabilité des actions utilisateurs | UI perfectible |
| Vue détaillée pour les variations intéressante | Tableau rigide pour l'utilisateur sur la forme comme le contenu |
| Gère SV et fait le lien avec les données de SNV | |
| Gère les STR | … mais vue détaillée encore inadéquate |
| L'écran d'accueil des cas peut être une bonne inspiration | |
| Permet de commenter en local et global les variations, gènes, cas | |
| Permet de préciser la cause de rejet d'une variation | … mais de manière peu pratique |
| Possibilités d'enregistrer et partager des filtres | … mais les possibilités de filtrage restent limitées |
| Configuration apparemment facile en admin via un ensemble de lignes de commande | |
| Reporting inclu | … mais sous format HTML/PDF uniquement et non customisable |