Cutevariant
Cutevariant est un outil développé en Python par Sacha Schutz du CHRU de Brest pour l'affichage et le filtrage facile de VCF.
Note
Cutevariant est disponible directement via pypi pip install cutevariant, pour
Windows 64 bits,
ou directement built depuis son repo.
Nécessite sqlite ≥ 3.32
Présentation générale
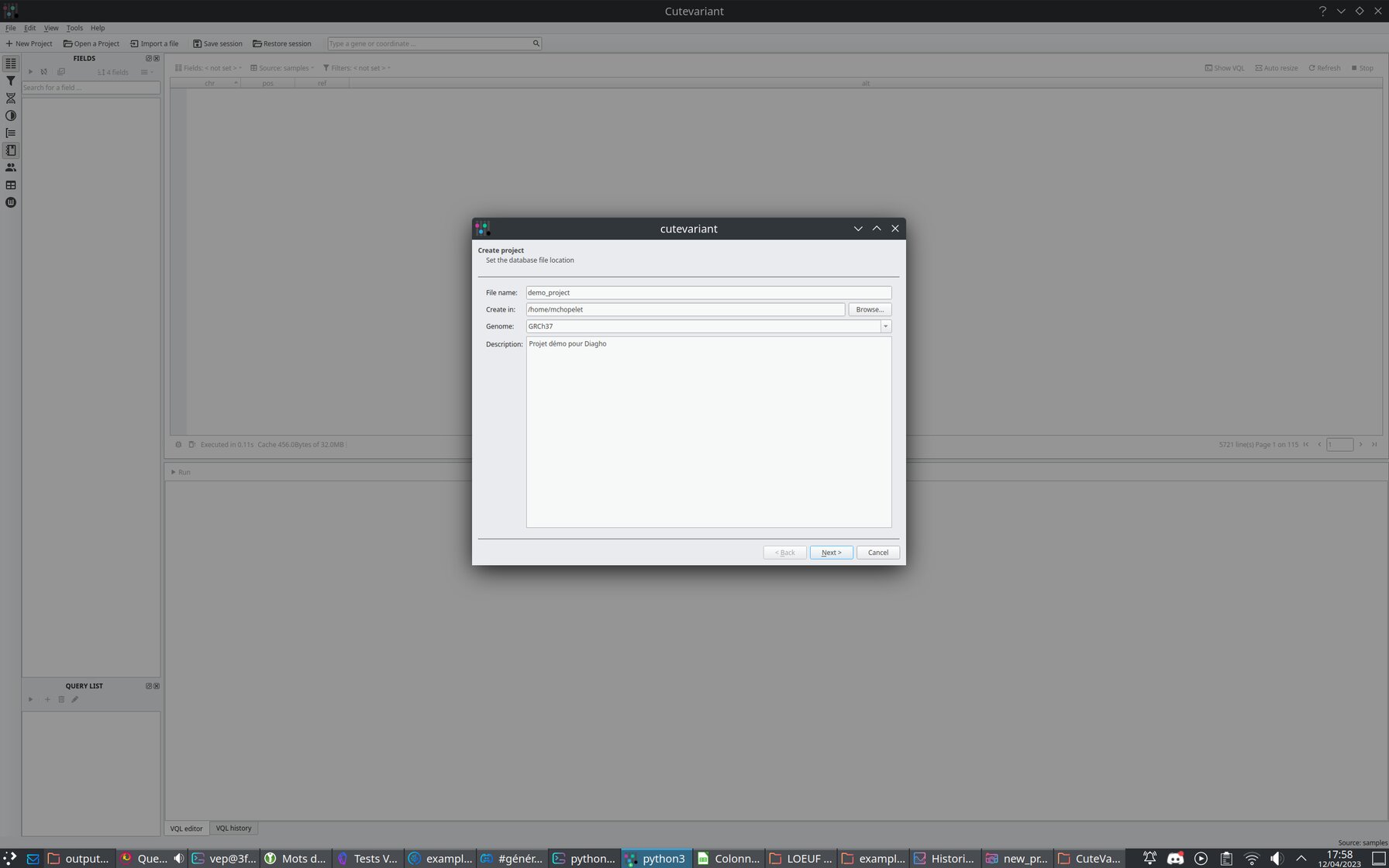
La première étape au lancement de Cutevariant est de créer un projet consistant en une base SQLite.
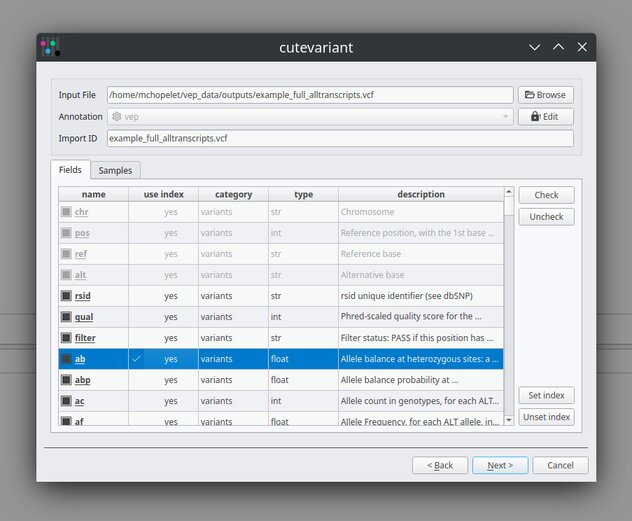
Cutevariant a une approche agnostique permettant le chargement de VCF annotés sous la
forme clef=valeur et respectant le standard VEP/SnpEff (gérant le multi-transcrits).
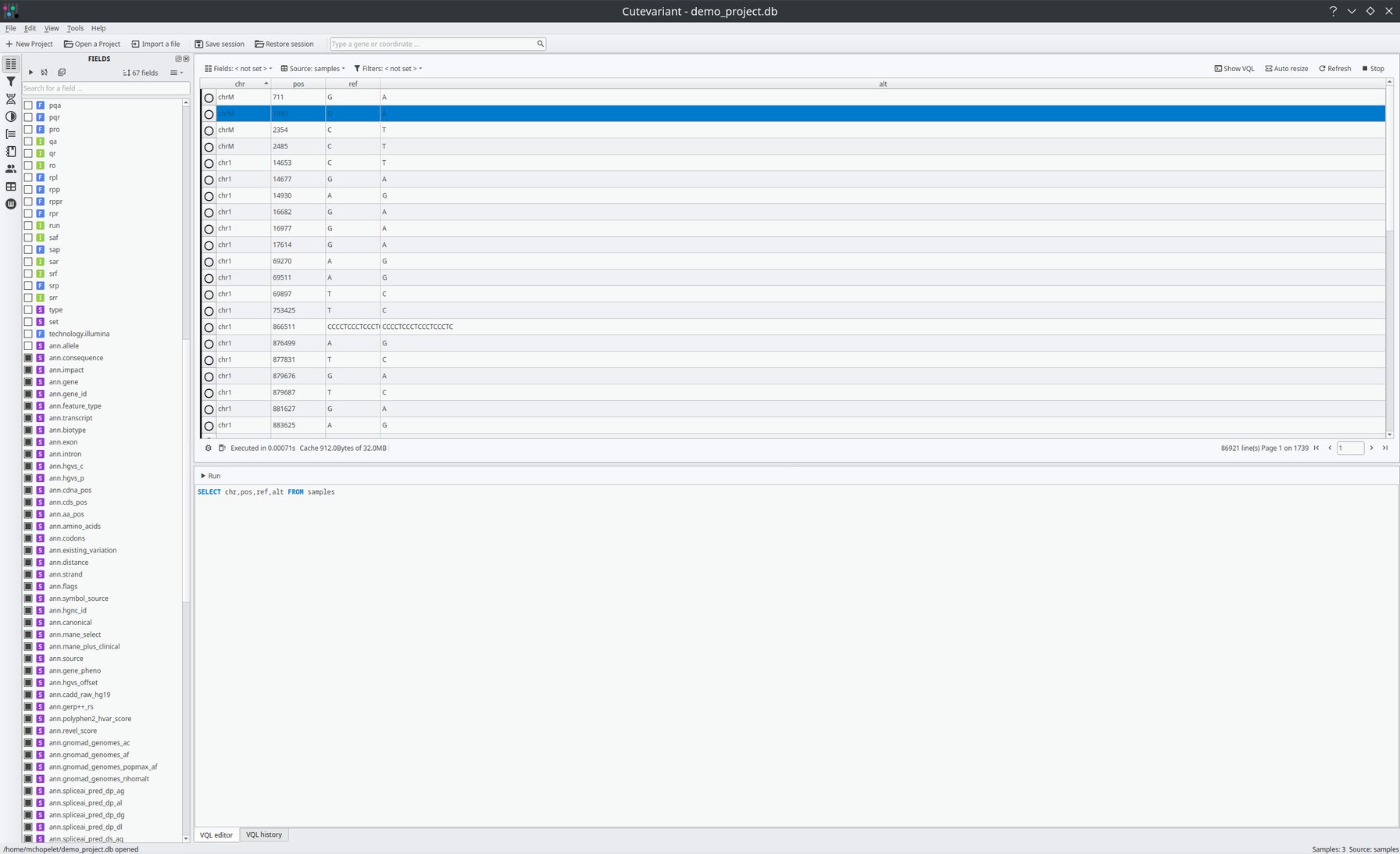
La gestion des colonnes, des filtres, des classifications peut se faire par le GUI tout comme en saisissant directement une requête VQL (équivalent de SQL).
C'est en l'occurrence un outil très bien réfléchi sur beaucoup d'aspects et digne de servir d'inspiration en divers points.
Interface
Permet d'avoir facilement le nombre de variations pour chaque valeur de chaque colonne. Peut servir de condition de filtrage en un double clic.

Permet de facilement catégoriser un ensemble de variations répondant à certaines conditions. Même fonctionnement des tags pour le prototype du Croisic.


Avis
| Points positifs | Points négatifs |
|---|---|
| Très simple à mettre en place | … mais client ne le rendant pas compatible pour une utilisation partagée |
| Approche totalement agnostique sur les VCF acceptés | |
| Parsing extrêmement rapide (5.8M var peu annotées chargées + indexées en ~13min) | … mais DB "jetables" n'ayant pas vocation à stocker des milliers d'exomes |
| Liberté totale sur les filtres, via GUI et requête DB | … mais le VQL rebute souvent les biologistes |
| Liberté totale sur les colonnes et sauvegarde de ces présets | … sauf la gestion de width qui est associée à une position et non un champ |
| Repose sur un système de plugins Qt permettant d'ajouter des controlleurs au besoin |
Notes additionnelles
C'est l'outil initialement retenu par Auragen dans le cadre du PFMG2025 bien que non adapté pour une utilisation dans une instance partagée.